吴恩达神经网络1-2-2_图神经网络进行药物发现-第1部分
吴恩达神经网络1-2-2
预测溶解度 (Predicting Solubility)
相关资料 (Related Material)
Jupyter Notebook for the article
Jupyter Notebook的文章
Drug Discovery with Graph Neural Networks — part 2
图神经网络进行药物发现-第2部分
Introduction to Cheminformatics
化学信息学导论
Deep learning on graphs: successes, challenges, and next steps (article by prof Michael Bronstein)
图上的深度学习:成功,挑战和下一步 (迈克尔·布朗斯坦教授的文章)
Towards Explainable Graph Neural Networks
走向可解释的图形神经网络
目录 (Table of Contents)
- Introduction介绍
- A Special Chemistry Between Drug Development and Machine Learning药物开发与机器学习之间的特殊化学
- Why Molecular Solubility is Important为什么分子溶解度很重要
- Approaching the Problem with Graph Neural Networks图神经网络解决问题
- Hands-on Part with DeepchemDeepchem的动手部分
- About Me关于我
介绍 (Introduction)
This article is a mix of theory behind drug discovery, graph neural networks and a practical part of Deepchem library. The first part will discuss potential applications of machine learning in drug development and then explain what molecular features might prove useful for the graph neural network model. We then dive into coding part and create a GNN model that can predict the solubility of a molecule. Let’s get started!
本文综合了药物发现,图形神经网络和Deepchem库的实际部分的理论知识。 第一部分将讨论机器学习在药物开发中的潜在应用,然后解释什么分子特征可能对图神经网络模型有用。 然后,我们深入编码部分,并创建可以预测分子溶解度的GNN模型。 让我们开始吧!
药物开发与机器学习之间的特殊化学 (A Special Chemistry between Drug Development and Machine Learning)
Drug development is a time-consuming process which might take decades to approve the final version of the drug [1]. It starts from the initial stage of drug discovery where it identifies certain groups of molecules that are likely to become a drug. Then, it goes through several steps to eliminate unsuitable molecules and finally tests them in real life. Important features that we look at during the drug discovery stage are ADME (Absorption, Distribution, Metabolism, and Excretion) properties. We can say that drug discovery is an optimization problem where we predict the ADME properties and choose those molecules that might increase the likelihood of developing a safe drug [2]. Highly efficient computational methods that find molecules with desirable properties speed up the drug development process and give a competitive advantage over other R&D companies.
药物开发是一个耗时的过程,可能需要数十年才能批准该药物的最终版本[1]。 它从药物发现的初始阶段开始,在此阶段它可以识别可能成为药物的某些分子组。 然后,它通过几个步骤来消除不合适的分子,并最终在现实生活中对其进行测试。 重要的特征,我们在药物开发阶段看是ADME(A bsorption,d istribution, 男 etabolism和E xcretion)性能。 可以说,药物发现是一个优化问题,我们可以预测ADME特性并选择可能增加开发安全药物可能性的分子[2]。 查找具有所需特性的分子的高效计算方法可加快药物开发过程,并提供优于其他研发公司的竞争优势。
It was only a matter of time before machine learning was applied to the drug discovery. This allowed to process molecular datasets with a speed and precision that had not been seen before [3]. However, to make the molecular structures applicable to machine learning, many complicated preprocessing steps have to be performed such as converting 3D molecular structures to 1D fingerprint vectors, or extracting numerical features from specific atoms in a molecule.
将机器学习应用于药物发现只是时间问题。 这样可以以前所未有的速度和精度处理分子数据集[3]。 但是,要使分子结构适用于机器学习,必须执行许多复杂的预处理步骤,例如将3D分子结构转换为1D指纹矢量 ,或从分子中的特定原子提取数值特征。
为什么分子溶解度很重要 (Why Molecular Solubility is Important)
One of the ADME properties, absorption, determines whether the drug can reach efficiently the patient’s bloodstream. One of the factors behind the absorption is aqueous solubility, i.e. whether a certain substance is soluble in water. If we are able to predict the solubility, we can also get a good indication of the absorption property of the drug.
ADME的特性之一就是吸收,它决定药物是否可以有效地到达患者的血液中。 吸收背后的因素之一是水溶性,即某种物质是否可溶于水。 如果我们能够预测溶解度,我们也可以很好地表明药物的吸收特性。
图神经网络解决问题 (Approaching the Problem with Graph Neural Networks)
To apply GNNs to molecular structures, we must transform the molecule into a numerical representation that can be understood by the model. It is a rather complicated step and it will vary depending on the specific architecture of the GNN model. Fortunately, most of that preprocessing is covered by external libraries such as Deepchem or RDKit.
要将GNN应用于分子结构,我们必须将分子转换为模型可以理解的数字表示形式。 这是一个相当复杂的步骤,并且会根据GNN模型的特定架构而有所不同。 幸运的是,大多数预处理都被Deepchem或RDKit之类的外部库所覆盖。
Here, I will quickly explain the most common approaches to preprocess a molecular structure.
在这里,我将快速解释预处理分子结构的最常用方法。
微笑 (SMILES)
SMILES is a string representation of the 2D structure of the molecule. It maps any molecule to a special string that is (usually) unique and can be mapped back to the 2D structure. Sometimes, different molecules can be mapped to the same SMILES string which might decrease the performance of the model.
SMILES是分子的2D结构的字符串表示。 它将任何分子映射到(通常)唯一且可以映射回2D结构的特殊字符串。 有时,不同的分子可以映射到相同的SMILES字符串,这可能会降低模型的性能。
指纹识别 (Fingerprints)

Fingerprints is a binary vector where each bit represents whether a certain substructure of the molecule is present or not. It is usually quite long and might fail to incorporate some structural information such as chirality.
指纹是一个二进制向量,其中每个位代表是否存在该分子的某个子结构 。 它通常很长,可能无法合并一些结构信息,例如手性 。
邻接矩阵和特征向量 (Adjacency Matrix and Feature Vectors)
Another way to preprocess a molecular structure is to create an adjacency matrix. The adjacency matrix contains information about the connectivity of atoms, where “1” means that there is a connection between them and “0” that there is none. The adjacency matrix is sparse and is often quite big which might not be very efficient to work with.
预处理分子结构的另一种方法是创建邻接矩阵 。 邻接矩阵包含有关原子连接性的信息,其中“ 1”表示原子之间存在连接,而“ 0”表示不存在连接。 邻接矩阵是稀疏的,并且通常很大,使用它可能不是很有效。

Together with this matrix, we can provide to the GNN model information about each individual atom and information about neighbouring atoms in a form of a vector. In the feature vector for each atom, there can be information about the atomic number, number of valence electrons, or number of single bonds. There is of course many more and they can fortunately be generated by RDKit and Deepchem,
与此矩阵一起,我们可以以矢量的形式向GNN模型提供有关每个单个原子的信息以及有关相邻原子的信息。 在每个原子的特征向量中,可以包含有关原子序数,价电子数或单键数的信息。 当然还有更多,它们可以由RDKit和Deepchem生成,

溶解度 (Solubility)
The variable that we are going to predict is called cLogP and is also known as octanol-water partition coefficient. Basically, the lower is the value the more soluble it is in water. clogP is a log ratio so the values range from -3 to 7 [6].
我们将要预测的变量称为cLogP和 也称为辛醇-水分配系数。 基本上,该值越低,它在水中的溶解度越高。 clogP是对数比率,因此值的范围是-3到7 [6]。
There is also a more general equation describing the solubility logS:
还有一个更通用的方程式描述溶解度logS :
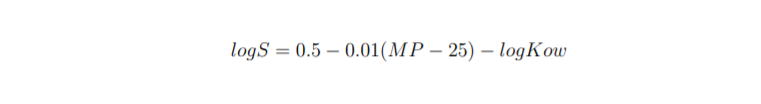
The problem with that equation is that MP is very difficult to predict from the chemical structure of the molecule [7]. All available solubility datasets contain only cLogP value and this is the value that we are going to predict as well.
该方程式的问题在于, 很难通过分子的化学结构来预测MP [7]。 所有可用的溶解度数据集仅包含cLogP值,这也是我们将要预测的值。
Deepchem的动手部分 (Hands-on Part with Deepchem)
Colab notebook that you can run by yourself is here.
您可以自己运行的Colab笔记本在这里。
Deepchem is a deep learning library for life sciences that is built upon few packages such as Tensorflow, Numpy, or RDKit. For molecular data, it provides convenient functionality such as data loaders, data splitters, featurizers, metrics, or GNN models. From my experience, it is quite troublesome to setup so I would recommend running it on the Colab notebook that I’ve provided. Let’s get started!
Deepchem是用于生命科学的深度学习库,它建立在Tensorflow,Numpy或RDKit等少数软件包的基础上。 对于分子数据,它提供了方便的功能,例如数据加载器,数据拆分器,特征化器,度量或GNN模型。 根据我的经验,设置起来很麻烦,所以我建议在我提供的Colab笔记本上运行它。 让我们开始吧!
Firstly, we will download a Delaney dataset, which is considered as a benchmark for solubility prediction task. We then load the dataset using CSVLoader class and specify a column with cLogP data which is passed into tasks argument. In smiles_field, name of the column with SMILES string have to be specified. We choose a ConvMolFeaturizer which will create input features in a format required by the GNN model that we are going to use.
首先,我们将下载Delaney数据集,该数据集被视为溶解度预测任务的基准。 然后,我们使用CSVLoader类加载数据集,并指定包含cLogP数据的列,该列将传递到task参数。 在smiles_field中,必须指定带有SMILES字符串的列的名称。 我们选择一个ConvMolFeaturizer,它将以我们将要使用的GNN模型所需的格式创建输入要素。
# Getting the delaney dataset
!wget https://raw.githubusercontent.com/deepchem/deepchem/master/datasets/delaney-processed.csv
from deepchem.utils.save import load_from_disk
dataset_file= "delaney-processed.csv"# Loading the data from the CSV file
loader = deepchem.data.CSVLoader(tasks=["ESOL predicted log solubility in mols per litre"], smiles_field="smiles", featurizer=deepchem.feat.ConvMolFeaturizer())
# Featurizing the dataset with ConvMolFeaturizer
dataset = loader.featurize(dataset_file)Later, we split the dataset using RandomSplitter and divide data into training and validation set. We also use a normalization for y values so they have zero mean and unit standard deviation.
之后,我们使用RandomSplitter分割数据集,并将数据分为训练和验证集。 我们还对y值使用归一化,因此它们的均值和单位标准差为零。
# Splitter splits the dataset # In this case it's is an equivalent of train_test_split from sklearnsplitter = deepchem.splits.RandomSplitter()# frac_test is 0.01 because we only use a train and valid as an exampletrain, valid, _ = splitter.train_valid_test_split(dataset,frac_train=0.7,frac_valid=0.29,frac_test=0.01)# Normalizer will normalize y values in the datasetnormalizer = deepchem.trans.NormalizationTransformer(transform_y=True, dataset=train, move_mean=True)train = normalizer.transform(train)test = normalizer.transform(valid)In this example, we will use a GraphConvModel as our GNN models. It’s an architecture that was created by Duvenaud, et al. You can find their paper here. There are other GNN models as a part of the Deepchem package such as WeaveModel, or DAGModel. You can find a full list of the models with required featurizers here.
在此示例中,我们将使用GraphConvModel作为我们的GNN模型。 这是Duvenaud等人创建的架构。 您可以在这里找到他们的论文。 Deepchem软件包中还包含其他GNN模型,例如WeaveModel或DAGModel。 您可以在此处找到具有所需功能的所有型号的完整列表。
In this code snippet, a person R2 score is also defined. Simply speaking, the closer this value is to 1, the better is the model.
在此代码段中,还定义了人员R2分数。 简单地说,该值越接近1,模型越好。
# GraphConvModel is a GNN model based on
# Duvenaud, David K., et al. "Convolutional networks on graphs for
# learning molecular fingerprints."
from deepchem.models import GraphConvModel
graph_conv = GraphConvModel(1,batch_size=50,mode="regression")
# Defining metric. Closer to 1 is better
metric = deepchem.metrics.Metric(deepchem.metrics.pearson_r2_score)Deepchem models use Keras API. The graph_conv model is trained with the fit() function. Here you can also specify the number of epochs. We get the scores with evaluate() function. Normalizer has to be passed here because y values need to be mapped again to the previous range before computing the metric score.
Deepchem模型使用Keras API。 graph_conv模型是使用fit()函数训练的。 在这里,您还可以指定时期数。 我们使用评价()函数获得分数。 必须在此处传递规范化器,因为在计算指标得分之前, y值需要再次映射到先前的范围。
# Fitting the model
graph_conv.fit(train, nb_epoch=10)# Reversing the transformation and getting the metric scores on 2 datasets
train_scores = graph_conv.evaluate(train, [metric], [normalizer])
valid_scores = graph_conv.evaluate(valid, [metric], [normalizer])And that’s all! You can do much more interesting stuff with Deepchem. They created some tutorials to show what else you can do with it. I highly suggest looking over it. You can find them here.
就这样! 您可以使用Deepchem做更多有趣的事情。 他们创建了一些教程来展示您还可以做什么。 我强烈建议您仔细检查一下。 您可以在这里找到它们。
Thank you for reading the article, I hope it was useful for you!
感谢您阅读本文,希望对您有所帮助!
关于我 (About Me)
I am an MSc Artificial Intelligence student at the University of Amsterdam. In my spare time, you can find me fiddling with data or debugging my deep learning model (I swear it worked!). I also like hiking :)
我是阿姆斯特丹大学的人工智能硕士研究生。 在业余时间,您会发现我不喜欢数据或调试我的深度学习模型(我发誓它能工作!)。 我也喜欢远足:)
Here are my social media profiles, if you want to stay in touch with my latest articles and other useful content:
如果您想与我的最新文章和其他有用内容保持联系,这是我的社交媒体个人资料:
Medium
中
Linkedin
领英
Github
Github
Personal Website
个人网站
翻译自: https://towardsdatascience.com/drug-discovery-with-graph-neural-networks-part-1-1011713185eb
吴恩达神经网络1-2-2
http://www.taodudu.cc/news/show-997428.html
相关文章:
- python 数据框缺失值_Python:处理数据框中的缺失值
- 外星人图像和外星人太空船_卫星图像:来自太空的见解
- 棒棒糖 宏_棒棒糖图表
- nlp自然语言处理_不要被NLP Research淹没
- 时间序列预测 预测时间段_应用时间序列预测:美国住宅
- 经验主义 保守主义_为什么我们需要行动主义-始终如此。
- python机器学习预测_使用Python和机器学习预测未来的股市趋势
- knn 机器学习_机器学习:通过预测意大利葡萄酒的品种来观察KNN的工作方式
- python 实现分步累加_Python网页爬取分步指南
- 用于MLOps的MLflow简介第1部分:Anaconda环境
- pymc3 贝叶斯线性回归_使用PyMC3估计的贝叶斯推理能力
- 朴素贝叶斯实现分类_关于朴素贝叶斯分类及其实现的简短教程
- vray阴天室内_阴天有话:第1部分
- 机器人的动力学和动力学联系_通过机器学习了解幸福动力学(第2部分)
- 大样品随机双盲测试_训练和测试样品生成
- 从数据角度探索在新加坡的非法毒品
- python 重启内核_Python从零开始的内核回归
- 回归分析中自变量共线性_具有大特征空间的回归分析中的变量选择
- python 面试问题_值得阅读的30个Python面试问题
- 机器学习模型 非线性模型_机器学习:通过预测菲亚特500的价格来观察线性模型的工作原理...
- pytorch深度学习_深度学习和PyTorch的推荐系统实施
- 数据库课程设计结论_结论:
- 网页缩放与窗口缩放_功能缩放—不同的Scikit-Learn缩放器的效果:深入研究
- 未越狱设备提取数据_从三星设备中提取健康数据
- 分词消除歧义_角色标题消除歧义
- 在加利福尼亚州投资于新餐馆:一种数据驱动的方法
- 近似算法的近似率_选择最佳近似最近算法的数据科学家指南
- 在Python中使用Seaborn和WordCloud可视化YouTube视频
- 数据结构入门最佳书籍_最佳数据科学书籍
- 多重插补 均值插补_Feature Engineering Part-1均值/中位数插补。
吴恩达神经网络1-2-2_图神经网络进行药物发现-第1部分相关推荐
- 吴恩达.深度学习系列-C4卷积神经网络-W2深度卷积模型案例
吴恩达.深度学习系列-C4卷积神经网络-W2深度卷积模型案例 (本笔记部分内容直接引用redstone的笔记http://redstonewill.com/1240/.原文整理的非常好,引入并添加我自 ...
- 吴恩达深度学习思维导图--来自刚入门的学生的自制版
吴恩达深度学习思维导图 学习吴恩达老师的课一学期了,在课程老师的要求下每周视频课都需要做思维导图,最后做了一份汇总版本的思维导图,与各位共享交流一下. 思维导图内的图片和部分内容是结合吴恩达老师的视频 ...
- 干货|吴恩达Coursera课程教你学习神经网络!
吴恩达Coursera机器学习课程系列笔记讲解 课程笔记|吴恩达Coursera机器学习 Week1 笔记-机器学习基础 干货|机器学习零基础?不要怕,吴恩达机器学习课程笔记2-多元线性回归 干货|机 ...
- 吴恩达深度学习之四《卷积神经网络》学习笔记
一.卷积神经网络 1.1 计算机视觉 举了几个例子,可以完成什么样的任务 最重要的是特征向量太大了,比如分辨率1000 x 1000 的彩色图片,三个颜色通道,维数是 3000000 意味着隐藏层第一 ...
- 吴恩达深度学习 | (12) 改善深层神经网络专项课程第三周学习笔记
课程视频 第三周PPT汇总 吴恩达深度学习专项课程共分为五个部分,本篇博客将介绍第二部分改善深层神经网络专项的第三周课程:超参数调试.Batch Normalization和深度学习框架. 目录 1. ...
- 吴恩达深度学习笔记- lesson4 卷积神经网络
文章目录 Week 1 卷积神经网络基础 4.1.1 计算机视觉(Computer vision) 4.1.2 边缘检测示例(Edge detection example) 4.1.3 更多边缘检测内 ...
- 从李飞飞、吴恩达、安德鲁的年度总结中,我们发现了三条2018年AI行业发展趋势...
"AI梦想远大,但它还只是一门年轻的科学.只有在深思熟虑.兼容并包的探索下,2018的AI才会取得更多的进步."李飞飞在其年终总结的最后写到. 首先,祝大家圣诞节快乐!(当然如果你 ...
- 吴恩达深度学习 —— 3.3 计算神经网络的输出
如图是一个两层的神经网络,让我们更深入地了解神经网络到底在计算什么. 我们之前说过逻辑回归,下图中的圆圈代表了回归计算的两个步骤,首先按步骤计算出z,然后在第二部计算激活函数,就是函数sigmoid( ...
- 吴恩达深度学习笔记2-Course1-Week2【神经网络基础:损失函数、梯度下降】
神经网络基础:损失函数.梯度下降 本篇以最简单的多个输入一个输出的1层神经网络为例,使用logistic regression讲解了神经网络的前向反向计算(forward/backward propa ...
- 干货|吴恩达Coursera课程教你学习神经网络二!
上一周的课程中讲了神经网络的结构以及正向传播(feed forward)过程,了解了神经网络是如何进行预测的,但是预测的结果怎么和真是结果进行比较以及发现了错误如何修改还没有提及. 这一周的课程中,介 ...
最新文章
- P2774 方格取数问题 网络最大流 割
- 《巨富们给年轻人的45个忠告》读后感
- VS2010-2015对C++11/14/17特性的支持
- CentOS 7 下安装 mysql ,以及用到的命令
- CodeForces - 932D Tree(树上倍增,好题)
- Spring Cloud Alibaba迁移指南(三):极简的 Config 1
- gitlab clone需要密码_搭建gitlab服务器最详教程
- 西瓜书+实战+吴恩达机器学习(十六)半监督学习(半监督SVM、半监督k-means、协同训练算法)
- [zsh] restart a zsh process
- Material Design之RecyclerView的使用(一)
- 如何完全卸载OneDrive (Windows 10 64bit)
- 阶段3 1.Mybatis_07.Mybatis的连接池及事务_1 今日课程内容介绍
- jeecg字典表-系统字典
- 一文带你了解手机运营商类api接口
- C语言编程方法技巧,C语言编程小技巧分享
- IDEA 打包docker镜像详解
- android 如何把图片设置成圆,Android 设置圆形图片 设置圆角图片
- VO快速搜索 宝马VO 宝马VO翻译 VO码查询工具
- 84行C++代码教你实现洛谷占卜功能
- 哈工大c语言作业,哈工大c语言-练习题
热门文章
- 计算机考研英语词汇书,求助:有知道电脑背考研英语单词的
- gcc是java的什么意思_为什么gcc支持Java而不是C#
- 通过Ajax方式上传文件(input file),使用FormData进行Ajax请求
- Entity Framework Logging and Intercepting Database Operations (EF6 Onwards)
- 【BZOJ4653】[Noi2016]区间 双指针法+线段树
- 玩转CSS3(一)----CSS3实现页面布局
- 利用 Docker 搭建单机的 Cloudera CDH 以及使用实践
- 使用了JDK自带的jconsole查看Tomcat运行情况
- RUNOOB python练习题31 根据已输入的字符判断星期几
- 漏洞发布平台-安百科技
