TCGA数据库的利用(三)—做差异分析的三种方法
今天更新TCGA数据库的利用系列第三篇文章,在对TCGA数据进行挖掘时,通常会筛选出来一些表达量显著异常的基因,作为后续研究的对象,这个筛选过程叫做差异分析;本篇文章将分为三大模块对差异分析进行介绍
关于差异分析的官方解释:
差异分析就是将一组资料的总变动量,依可能造成变动的因素分解成不同的部份,并且以假设检定的方法来判断这些因素是否确实能解释资料的变动。
我自己的一点理解:差异分析就是对总体样本数据中非正常数据的筛选。对于转录组数据进行差异分析时,limma 、edgeR、DESeq2这三种程序包都可以(limma相对较受欢迎),大部分科研性文章基本上是用其中一种方法取筛选差异表达基因,但为了使得结果更加准确,在做毕业课题时我把三种方法都做了一遍,把它们结果的交集作为筛选出来的差异表达基因。
不管用那一种方法做差异分析,基本上要做的步骤就是:一,创建表达矩阵;二、创建设计矩阵(分组矩阵);三、得到差异表达分析矩阵。
但不同包基于算法、数据模型不同,所用的函数、筛选标准也大不相同,所以用代码实现时结果有很大的差别。
无论用那种包做差异分析,在做之前必须要保证需要用到的包已经安装成功。在R语言中安装程序包的代码(其中的一种方式)如下:
source('https://bioconductor.org/biocLite.R')#加载的网址都一样biocLite("edgeR")#把双引号的内容换成你所需要程序包的名称即可limma做差异分析
传入原始样本基因表达矩阵(表达矩阵格式如下图)
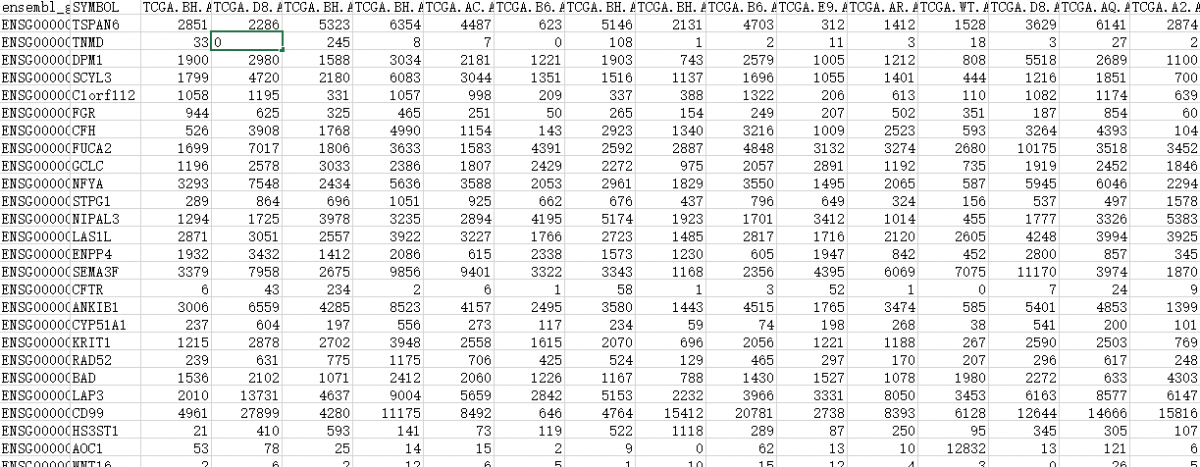
接下来就是对基因表达矩阵进行一些处理,让样本名变成数据矩阵的列名,基因名变成数据矩阵的行名,同时把ensembl_symbl那一列去掉(用express_rec <- express_rec[,(-1)]命令即可),变成如下这个格式:
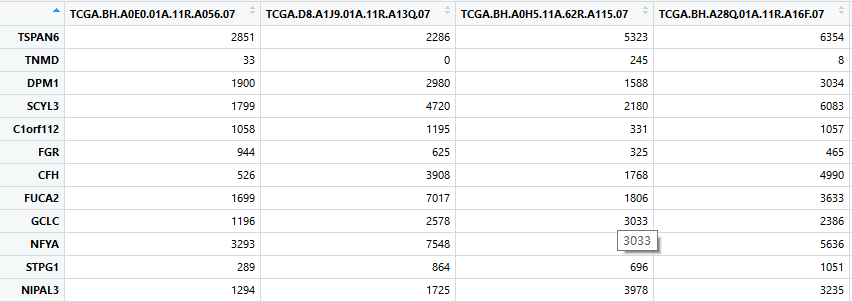
表达矩阵里面的数据太大,但为了使数据呈现正态分布,需要对数据进行标准化,这里我用的是函数log(express_rec,2),在标准化之前,需要把表达矩阵内为0的数据赋值为1,目的是为了防止取log时,数据变为负无穷大。
(express_rec[express_rec==0<-1)下面进行分组矩阵的组建,首先提前创建好一个矩阵列表,如下,行名为样本编码,列名为样本类型,如下面这种格式:
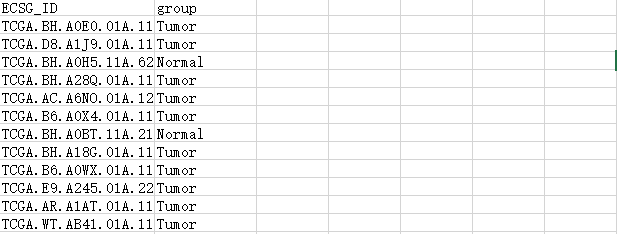
而limma包用到的设计矩阵是下面这种格式:
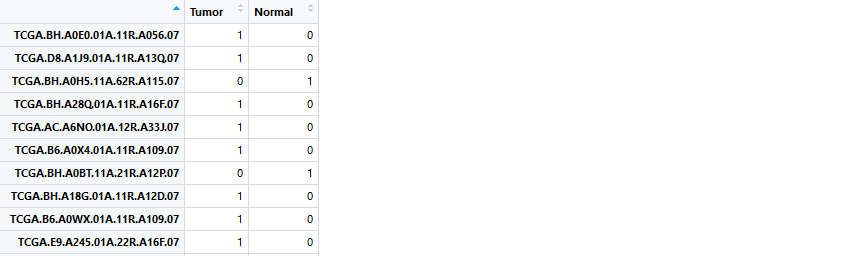
实现代码如下:
rownames(group_text) <- group_text[,1]group_text <- group_text[c(-1)]Group <- factor(group_text$group,levels = c('Tumor','Normal'))design <- model.matrix(~0+Group)colnames(design) <- c('Tumor','Normal')rownames(design) <- rownames(group_text)接下来的步骤依次进行数据拟合、经验贝叶斯检验、筛选差异表达基因
fit <- lmFit(express_rec,design)#制作比对标准;contrast.matrix <- makeContrasts(Tumor - Normal,levels=design)fit2 <- contrasts.fit(fit,contrast.matrix)#进行经验贝叶斯检验;fit2 <- eBayes(fit2)#基于logFC为标准,设置数量上限为30000,调整方法为fdr;all_diff <- topTable(fit2, adjust.method = 'fdr',coef=1,p.value = 1,lfc <- log(1,2),number = 30000,sort.by = 'logFC')#从高到低排名;limma包的另一种方法,精确权重法(voom),然后把筛选得到的差异表达基因写入csv文件中。
#limma的另一种方法;dge <- DGEList(counts = express_rec)dge <- calcNormFactors(dge)#表达矩阵进行标准化;v <- voom(dge, design,plot=TRUE)#利用limma_voom方法进行差异分析;fit <- lmFit(v, design)#对数据进行线性拟合;fit <- eBayes(fit)#贝叶斯算法组建all <- topTable(fit, coef=ncol(design),n=Inf)#从高到低排名;sig.limma <- subset(all_diff,abs(all$logFC)>1.5&all$P.Value<0.05)#进行差异基因筛选;write.csv(sig.limma,'C:/Users/FREEDOM/Desktop/TCGA_data/limm_diff.csv')#写入csv文件中;DESeq2做差异分析
第一步跟limma程序包一样,读入表达矩阵,对表达矩阵进行数据处理
express_rec<- read.csv('C:/Users/FREEDOM/Desktop/TCGA_data/after_note2.csv')#读取数据group_text <- read.csv('C:/Users/FREEDOM/Desktop/TCGA_data/group_text.csv')library('DESeq2')#加载包;install.packages('rpart')#含有这个包可忽略,没有的时候才安装;express_rec <- express_rec[,-1]rownames(express_rec) <-express_rec[,1]express_rec <- express_rec[(-1)]#表达矩阵的处理;rownames(group_text) <- group_text[,1]创建分组矩阵
rownames(group_text) <- group_text[,1]group_text <- group_text[c(-1)]#分组矩阵的数据处理;all(rownames(group_text)==colnames(express_rec))#确保表达矩阵的列名与分组矩阵行名相一致;构建 DESeq2 所需的 DESeqDataSet 对象
dds <- DESeqDataSetFromMatrix(countData=express_rec, colData=group_text, design<- ~ group) #DESeq2的加载head(dds)dds <- dds[rowSums(counts(dds)) > 1, ] #过滤一些low count的数据;使用DESeq进行差异表达分析,返回 results可用的DESeqDataSet对象
> dds <- DESeq(dds)#DESeq进行标准化;estimating size factorsestimating dispersionsgene-wise dispersion estimatesmean-dispersion relationshipfinal dispersion estimatesfitting model and testing-- replacing outliers and refitting for 2819 genes-- DESeq argument 'minReplicatesForReplace' = 7 -- original counts are preserved in counts(dds)estimating dispersionsfitting model and testing可以用summary函数查看表达基因上下调分布基本情况
> summary(res)#查看经过标准化矩阵的基本情况;
out of 24823 with nonzero total read countadjusted p-value < 0.1LFC > 0 (up) : 7590, 31%LFC < 0 (down) : 5120, 21%outliers [1] : 0, 0%low counts [2] : 0, 0%(mean count < 0)[1] see 'cooksCutoff' argument of ?results[2] see 'independentFiltering' argument of ?results最后提取差异表达基因,存入csv文件中。
edgeR进行差异分析
这个包的操作步骤与limma包类似,首先就是读入数据,创建表达、分组矩阵。
path = 'C:/Users/FREEDOM/Desktop/TCGA_data/after_note2.csv'path1 ='C:/Users/FREEDOM/Desktop/TCGA_data/group_text.csv'express_rec <- read.csv(path,headers <- T)#读取表达矩阵;group_text <- read.csv(path1,headers <- T)#读取分组矩阵;library(edgeR)#加载edgeR包express_rec <- express_rec[,-1]rownames(express_rec) <- express_rec[,1]express_rec <- express_rec[(-1)]#创建表达矩阵;rownames(group_text) <- group_text[,1]group_text <- group_text[c(-1)]#加载分组矩阵;group <-factor(group_text$group)dge <- DGEList(counts = express_rec,group = group)#构建DEList对象;y <- calcNormFactors(dge)#利用calcNormFactor函数对DEList对象进行标准化(TMM算法) #创建设计矩阵,跟Limma包相似;Group <- factor(group_text$group,levels = c('Tumor','Normal'))design <- model.matrix(~0+Group)colnames(design) <- c('Tumor','Normal')rownames(design) <- rownames(group_text)#创建分组矩阵;edgeR包创建的分组矩阵与limma一样,是以factor因子格式展现出来

接下来依次进行构建DGEList对象、利用TMM算法对数据进行标准化、估计离散值、数据拟合、创建对比矩阵、对数据做QL-text检验、差异表达基因写入csv文件中
dge <- DGEList(counts = express_rec,group = group)#构建DEList对象;y <- calcNormFactors(dge)#利用calcNormFactor函数对DEList对象进行标准化(TMM算法) y <- estimateDisp(y,design)#估计离散值(Dispersion)fit <- glmQLFit(y, design, robust=TRUE)#进一步通过quasi-likelihood (QL)拟合NB模型TU.vs.NO <- makeContrasts(Tumor-Normal, levels=design)#这一步主要构建比较矩阵;res <- glmQLFTest(fit, contrast=TU.vs.NO)#用QL F-test进行检验# ig.edger <- res$table[p.adjust(res$table$PValue, method = "BH") < 0.01, ]#利用‘BH’方法;result_diff <- res$table#取出最终的差异基因;write.csv(edge_diff,'C:/Users/FREEDOM/Desktop/TCGA_data/edgeR_diff2.csv')以上就是做差异分析三种R包的使用方法,关于本篇文章涉及到的完整源码的获取方式:关注公众号:小张Python,后台回复关键词:差异分析 即可。
TCGA数据库的利用(三)—做差异分析的三种方法相关推荐
- TCGA数据库的利用(二)—— 数据处理
上一篇文章介绍的是关于TCGA数据的下载,如果不太清楚怎么下载数据的读者请参考这篇文章:TCGA数据库的利用(一)-- 数据下载!,而本篇文章主要介绍一下数据的处理过程,因为数据下载之后都是单一样本存 ...
- java list 删除 遍历_Java list利用遍历进行删除操作3种方法解析
Java list利用遍历进行删除操作3种方法解析 这篇文章主要介绍了Java list利用遍历进行删除操作3种方法解析,文中通过示例代码介绍的非常详细,对大家的学习或者工作具有一定的参考学习价值,需 ...
- 修改mysql数据库默认字符集_MySQL数据库之修改mysql默认字符集的两种方法详细解析...
本文主要向大家介绍了MySQL数据库之修改mysql默认字符集的两种方法详细解析 ,通过具体的内容向大家展现,希望对大家学习MySQL数据库有所帮助. (1) 最简单的修改方法,就是修改mysql的m ...
- java 遍历删除list_Java list利用遍历进行删除操作3种方法解析
这篇文章主要介绍了Java list利用遍历进行删除操作3种方法解析,文中通过示例代码介绍的非常详细,对大家的学习或者工作具有一定的参考学习价值,需要的朋友可以参考下 Java三种遍历如何进行list ...
- 用matlab计算稳态误差,利用Matlab求稳态误差的两种方法.
利用Matlab求稳态误差的两种方法 摘要:稳态误差是系统控制精度或抗扰动能力的一种度量,它是稳态性能的一个重要指标.本文介绍利用Matlab的控制系统工具箱和Simulink工具箱求取系统误差稳态的 ...
- mysql数据库设置连接数_MySQL数据库之修改MYSQL最大连接数的3种方法分享
本文主要向大家介绍了MySQL数据库之修改MYSQL最大连接数的3种方法分享,通过具体的内容向大家展现,希望对大家学习MySQL数据库有所帮助. 方法一:进入MYSQL安装目录 打开MYSQL配置文件 ...
- dapper mysql 批量_MySQL数据库之c#mysql批量更新的两种方法
本文主要向大家介绍了MySQL数据库之c#mysql批量更新的两种方法 ,通过具体的内容向大家展现,希望对大家学习MySQL数据库有所帮助. 总体而言update 更新上传速度还是慢. 1: 简单的 ...
- mysql如何通过数据库修改root_MySQL数据库之MySQL——修改root密码的4种方法(以windows为例)...
本文主要向大家介绍了MySQL数据库之MySQL--修改root密码的4种方法(以windows为例) ,通过具体的内容向大家展现,希望对大家学习MySQL数据库有所帮助. 方法1: 用SET PAS ...
- 一元三次多项式因式分解的两种方法
参考文献: 张育波. 一元三次多项式因式分解的两种方法[J]. 初中数学教与学, 2007, No.160(04):42.
最新文章
- 亿级流量压力来袭,你的网站会被击垮吗?(上篇)
- IBM大裁70% 员工,撕掉了国内大批伪AI企业最后一块遮羞布!
- [洛谷P4626]一道水题 II
- 优秀的软件测试人员应该具备的素质
- [题解]BZOJ1004 序列函数
- SSL-练习题目:种树 题解
- 服务器如何用显示器更改ip,ip地址怎么改
- 5·19网络故障:DNS服务器被攻击
- iOS-使用代码约束布局(Masonry)
- 密码破解之Esxi重置root密码
- Atitit.android js 的键盘按键检测Back键Home键和Menu键事件
- react native+typescript创建移动端项目-(慕课网喜马拉雅项目笔记)-(二,导航器navigator)
- 2020 年百度之星·程序设计大赛 - 初赛二
- 吉林省吉林市谷歌高清卫星地图下载(百度网盘离线包下载)
- 8051单片机原理学习
- 听云-服务器监控,window+tomcat配置
- CSDN日报20170317——《转行穷三年?未必!》
- 解析自动休眠---实现自动关机
- php扩展cURL执行中途无响应
- 阿里巴巴高效的页面动画解决方案——Ant Motion Design
